Un team di ricercatori italiani ha sviluppato un software innovativo per anticipare le mosse del SARS-CoV-2. Questo strumento permette di prevedere quali varianti del virus prenderanno il sopravvento, facilitando lo sviluppo di vaccini e farmaci anti-Covid più efficaci e tempestivi.
Il progetto nasce da una collaborazione tra l’Azienda Ospedaliero-Universitaria Pisana (Aoup), il Politecnico di Milano e l’INMI “Lazzaro Spallanzani” IRCCS di Roma.
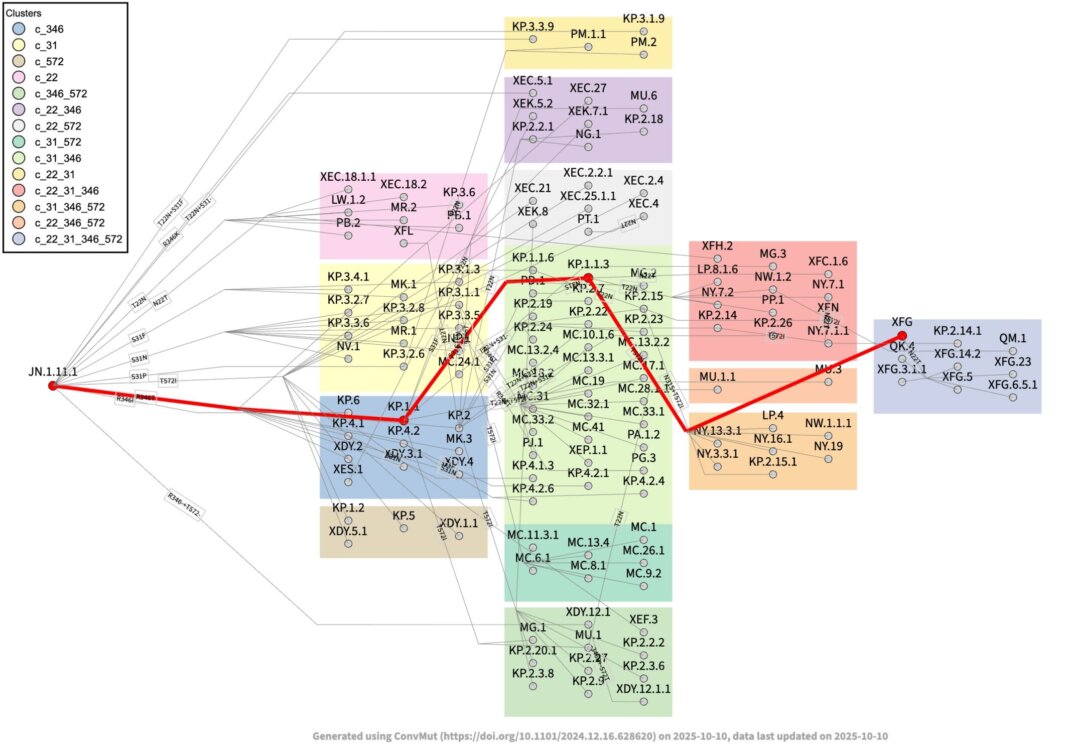
Il software, chiamato ConvMut, si basa su un affascinante fenomeno biologico: l’evoluzione convergente. In natura, specie anche molto diverse tra loro a volte sviluppano caratteristiche simili per adattarsi allo stesso ambiente. Allo stesso modo, il virus SARS-CoV-2 sviluppa ripetutamente le stesse mutazioni in diverse parti del mondo per diventare più contagioso. ConvMut è in grado di identificare proprio queste mutazioni “vincenti”.
L’idea è stata portata avanti da Daniele Focosi, ematologo e virologo dell’Aoup, insieme ai gruppi di lavoro di Fabrizio Maggi dello Spallanzani di Roma e di Anna Bernasconi del Politecnico di Milano.
Oggi la sfida principale contro il Covid-19 riguarda la protezione dei pazienti più fragili, come gli immunocompromessi. “Il virus continua a mutare per adattarsi,” spiega Daniele Focosi, “e questo richiede un aggiornamento annuale dei vaccini”. Il problema è che tra la scelta della variante da usare e la distribuzione del vaccino passano mesi. “Spesso accade che il vaccino aggiornato non riesca a tenere il passo dell’evoluzione del virus,” sottolinea Focosi.
Qui entra in gioco il nuovo software. “Prevedere con mesi di anticipo quale sarà il ceppo dominante ci permetterebbe di avere vaccini ancora più mirati“, aggiunge Fabrizio Maggi. ConvMut analizza le tendenze evolutive per capire quali mutazioni della proteina Spike, il bersaglio dei vaccini, diventeranno cruciali nei mesi a venire.
Il software automatizza un processo che prima richiedeva un’enorme cura manuale. Come spiega Anna Bernasconi, “ConvMut compila grafici di evoluzione convergente quasi in tempo reale, raggruppando i lignaggi virali in base alle mutazioni che hanno in comune”.
Per funzionare, lo strumento attinge a un database immenso: gli oltre 17 milioni di sequenze virali condivise sulla piattaforma internazionale GISAID, su cui ConvMut è ora liberamente accessibile a tutta la comunità scientifica.
Le applicazioni non si fermano ai vaccini. Il software sarà fondamentale anche per adattare gli anticorpi monoclonali, una delle terapie più importanti per i pazienti immunocompromessi. Prevedere l’evoluzione del virus aiuterà le aziende farmaceutiche a indirizzare meglio i loro sforzi, riducendo il rischio di sviluppare farmaci che diventano rapidamente inefficaci.